Initialising ...
Initialising ...
Initialising ...
Initialising ...
Initialising ...
Initialising ...
Initialising ...
松本 淳; 石田 恒
no journal, ,
近年、さまざまな状態にあるリボソームの3次元立体構造が、クライオ電子顕微鏡法と3次元再構成技術により解かれている。本研究では、これら多くのリボソームの3次元立体構造を、Elastic networkモデルの基準振動解析法とフィッティング手法を用いて解析する。われわれは、X線結晶構造解析により解かれたリボソームの原子分解能構造を、基準振動解析法によって変形し、変形構造のライブラリを構築した。その後、そのライブラリを探索し、リボソームの各EM構造にうまく当てはまる原子分解能構造を見つけ出した。その探索では、フィッティング手法を用いた。このようにして見つけ出された原子分解能構造を比較した結果、リボソームがラチェト様の構造変形を好むことを見いだした。
中川 洋; 城地 保昌*; 北尾 彰朗*; 片岡 幹雄
no journal, ,
Most globular proteins work in an aqueous milieu and water molecules located at the protein surface strongly affects the protein stability and function. Structure and dynamics of hydration water on the protein, staphylococcal nuclease, were examined at various hydration levels to study the hydration effect on dynamics. We found that the hydration dependent protein dynamical transition around 240 K shows the threshold hydration level between 0.30 and 0.42 (g water/g protein). Below the threshold, hydration water is localized to form several independent clusters on the surface, while above the threshold level the hydration water encircles the protein via hydrogen bond, suggesting the percolation transition. The MD simulations at various hydration indicate that the size of the cluster of hydrogen bonded hydration water dramatically increased above the threshold level of the hydration level. We concluded that the dynamics of hydration water is coupled with the protein dynamics via hydrogen-bond network on the protein-water interface. Such a dynamical coupling drives the hydration dependent protein dynamical transition.
村上 洋; 西 孝樹; 豊田 祐司
no journal, ,
蛋白質水溶液のテラヘルツ分光を含む赤外吸収分光は溶媒である水の吸収が非常に大きく蛋白質起因の信号を得ることが困難なために、凍結乾燥試料などが主な測定対象であった。発表者らは逆ミセルに水中蛋白質を導入することで、蛋白質起因のテラヘルツ吸収やアミドAバンドの測定を可能にした。逆ミセルのサイズは逆ミセル溶液中の水と界面活性剤濃度の比により決まるが、その制御は水和水など蛋白質の周りの環境が蛋白質の運動にどのような効果を与えるかを調べるために必要である。発表者らは蛋白質水溶液と界面活性剤を混合してペースト状にした後に脱水し、無極性溶媒に溶かすという、新しい蛋白質逆ミセル調製法を開発することにより、赤外分光可能な蛋白質濃度(10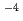 mol/l程度)以上の試料の逆ミセルサイズ制御に成功した。化学量論的モデルを用いた蛋白質逆ミセルの吸収スペクトルの解析により、蛋白質に水和する水の個数の定量化可能性が示された。
mol/l程度)以上の試料の逆ミセルサイズ制御に成功した。化学量論的モデルを用いた蛋白質逆ミセルの吸収スペクトルの解析により、蛋白質に水和する水の個数の定量化可能性が示された。
米谷 佳晃; 藤井 聡*; 皿井 明倫*; 河野 秀俊; 郷 信広
no journal, ,
Sequence dependence of DNA conformational deformability is one of important factors in the sequence-specific recognition by regulatory DNA-binding proteins. Intrinsic mechanical properties of DNA will make a dominant contribution, but the other effect of hydration cannot be neglected. A well known water spine is composed of highly ordered water molecules in the minor groove of specific DNA sequences. Emergence of such a hydration pattern depends on the sequence, raising the following question. How is the hydration related with the conformational properties of DNA? To address this question, we analyzed the deformability and hydration of various DNA sequences using the ensembles of DNA conformations generated by molecular dynamics simulations. The result showed a correlation between the DNA deformability and the hydration pattern. It was suggested that the obtained relation is associated with the coupled motions of DNA base-pair steps and hydration water.
樋口 真理子; Pinak, M.; 斎藤 公明
no journal, ,
複数のDNA損傷が集中して生じたものをクラスターDNA損傷と呼ぶ。これは放射線によって生じるDNA損傷の特徴でもある。クラスター損傷になると単独の損傷よりも修復されにくくなるが、その詳細な原因は不明である。具体的な例では、8oxoGの相補鎖の数ベースペア離れた位置に別の損傷があると、修復酵素MutMによる8oxoGの修復が阻害される。阻害率の高さは損傷の種類によって異なり、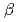 -
-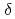 鎖切断は阻害率が高いが、
鎖切断は阻害率が高いが、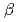 鎖切断はAPサイトと同程度である。本研究では、分子動力学シミュレーションを用いて、クラスター損傷を持つDNAの構造を調べた。用いたモデル構造は3種類で、それぞれ8oxoGの相補鎖の1ベースペア離れた位置に、(1)
鎖切断はAPサイトと同程度である。本研究では、分子動力学シミュレーションを用いて、クラスター損傷を持つDNAの構造を調べた。用いたモデル構造は3種類で、それぞれ8oxoGの相補鎖の1ベースペア離れた位置に、(1)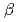 -
-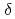 鎖切断,(2)
鎖切断,(2)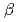 鎖切断,(3)APサイトを配置した。これらのモデルを用いたシミュレーション結果と実験データとの比較により、修復阻害率とDNA構造変化の関係について議論する。
鎖切断,(3)APサイトを配置した。これらのモデルを用いたシミュレーション結果と実験データとの比較により、修復阻害率とDNA構造変化の関係について議論する。
石田 恒
no journal, ,
Homologous recombination occurs in all organisms, allowing for genetic exchange, the reassortment of genes along chromosomes, and the repair of broken DNA strands. A four-stranded DNA structure called a Holliday junction (HJ) is a central intermediate state in homologous recombination, and one of the crucial steps in the process of recombination is branch migration of the HJ. In Escherichia coli, branch migration of the HJ is promoted by proteins RuvA and RuvB. To understand the relationship between the conformational changes in RuvB and the translocation of the HJ DNA, molecular dynamics (MD) simulations of the RuvA/RuvB/HJ DNA complex were carried out at a constant pressure of one bar and a temperature of 350 K. It was found that several low frequency modes showed the motions of the RuvB hexamer were coupled with the translational motion of DNA.
松本 富美子; Piroddi, N.*; Belus, A.*; Scellini, B.*; 前田 佳代*; Tesi, C.*; Poggesi, C.*; 前田 雄一郎*; 藤原 悟
no journal, ,
肥大型心筋症は遺伝性の心疾患で、心筋中のトロポニン(TnT, TnI, TnCの三量体から構成される)の遺伝性変異によって発症することが知られている。われわれは以前、TnT(E244D)とTnT(K247R)変異を交換導入した筋原繊維のATPase活性を測定し、これらの変異がカルシウム感受性を変えることなく筋肉の最大活性を増大させることを報告した。しかし、この現象と実際の筋肉収縮力の変化とを直接説明できずにいた。そこで本研究では、単一筋原繊維を用いて、これらの変異体が筋収縮力に及ぼす影響を直接測定した。その結果、張力の変化はATPase活性変化と全く同様の現象を示唆し、これらの変異体はカルシウム感受性に影響を与えずに最大張力を増大させるIII型心筋症と呼ばれる疾患を発症させることを明らかにした。また、これらの変異は、キネティクスには影響を与えないことを明らかにした。
藤原 悟; 小田 俊郎*; Plazanet, M.*; 松本 富美子
no journal, ,
F-アクチンは、真核細胞に最も豊富に存在し、細胞運動に関係する実に多様な機能を持つ。どのようにして、このような多様な機能が可能となるかを理解するためには、アクチン分子の内部運動から分子間の相対的運動、そしてF-アクチン全体の運動までのさまざまなレベルでの運動特性を明らかにすることが重要である。われわれは、そのためにF-アクチン及びアクチン単量体(G-アクチン)の中性子非干渉性弾性散乱実験を行い、F-アクチン及びG-アクチンの運動特性の違いを明らかにした。さらにアクチン分子内部の運動特性を詳しく特徴付けるため、中性子準弾性散乱実験を行った。第1水和層まで含む水和粉末試料及び「バルク」水まで含む試料についての測定を行った結果、F-アクチン及びG-アクチンのいずれも、少なくとも2種類の異なった振幅と速度を持つ運動が存在すること、そして含水量の増大は、これらの動きを速くさせること、そしてG-アクチンはF-アクチンよりも、より大きな振幅及び速い速度の運動を示す傾向があることを明らかにした。
徳久 淳師; 高 潤一郎; 森林 健悟; 乙部 智仁; 甲斐 健師; 中村 龍史; 福田 祐仁; 河野 秀俊; 郷 信広
no journal, ,
A promising tool for 3D-structural determination from a single-molecule without crystallized is X-ray free electron lasers (XFELs), which are now under construction in Europe, USA and Japan, independently. Our final goal is to develop a computational method for the determination of single-molecule 3D-strucures and to suggest feasible parameters of the target molecule and the XFEL device. When a molecule is irradiated by a coherent X-ray pulse with an ultrashort duration time and an extremely high intensity, a continuous diffraction pattern (2D speckle pattern) is observed. It is necessary to collect many 2D speckle patterns from different orientations of the molecule to obtain information sufficient for the structure determination. In the meeting, we present our strategy for determining 3D structures of biomolecules and suggest some critical parameters of the XFEL device and the target molecules required for realizing the 3D structure determination.
山口 繁生*; 上久保 裕生*; 栗原 和男; 黒木 良太; 新村 信雄*; 清水 伸隆*; 山崎 洋一*; 片岡 幹雄*
no journal, ,
Hydrogen bond dominates various physical properties in biological molecules and biological chemical reactions including proton transfer, hydrolysis, and so on. A special type of hydrogen bonds, the short hydrogen bond (SHB), has been proposed to have an important role in protein structure and function. The physical properties of SHBs, however, are still under debate. In the photoactive yellow protein (PYP), a light sensor protein, two SHBs near the chromophore have been revealed by X-ray crystallographic studies. The bond distances of the SHBs are modulated during the photo-reaction, resulting in the local proton transfer. From these facts, it can be postulated that these SHBs play a crucial role in the local proton transfer. Using high-resolution neutron crystallography in combination with high-resolution X-ray crystallography, we identified two deuterium atoms involved in two SHBs in PYP. One SHB, between the chromophore and E46, is identified as a low-barrier hydrogen bond (LBHB); the other, between the chromophore and Y42, is a short ionic hydrogen bond (SIHB). This is the first direct evidence of the coexistence of two distinct SHBs in the ground state of a protein. At the conference, we will present the neutron crystallographic structure and discuss the two SHBs in PYP.